Der Schweizerische Ziegenzuchtverband wechselt die Technologie für die Abstammungskontrolle auf die SNP-Typisierung. In den zurückliegenden Wochen wurden die Grundlagen für die SNP-basierte Abstammungskontrolle gelegt. SNP sind punktuelle Veränderungen im Erbgut. Dadurch eignen sie sich zur Charakterisierung und eindeutigen Identifikation von verschiedenen Tieren einer Population.
Abstammungskontrolle
Korrekte Abstammungen sind die Grundlage für sämtliche genetische Auswertungen (Zuchtwertschätzung) und damit für die Zucht im Allgemeinen. Daneben sind korrekte Abstammungen auch für das Monitoring von Populationen (Inzuchtentwicklung) wichtig.
Bisher wurde die Abstammung beim Schweizerischen Ziegenzuchtverband (SZZV) mittels «Mikrosatelliten» untersucht. Diese werden nun abgelöst durch die SNP-Analyse. Wie auch bei den Mikrosatelliten werden im Rahmen der Abstammungskontrolle die Ergebnisse aus der SNP-Analyse eines Tiers mit den Ergebnissen seinen Eltern verglichen.
Im Hintergrund stehen die Mendelschen Vererbungsregeln (siehe Box unten). In der SNP-basierten Abstammungskontrolle werden die SNPs gezählt, an denen die Mendelschen Regeln beim Vergleich Tier – Elter verletzt werden. Die Summe über alle SNPs ergibt das Ergebnis für die Abstammungskontrolle. Ein Schema mit 10 SNPs als Beispiel ist in Abbildung 1 zu sehen.
Routinemässig werden bei der Ziege 195 SNPs dafür verwendet. Grundsätzlich ist eine 100% Übereinstimmung das Ziel. Da jedoch auch technische Fehler möglich sind, werden auch Abstammungen mit 1-2 Fehlern auf SNP-Ebene akzeptiert.
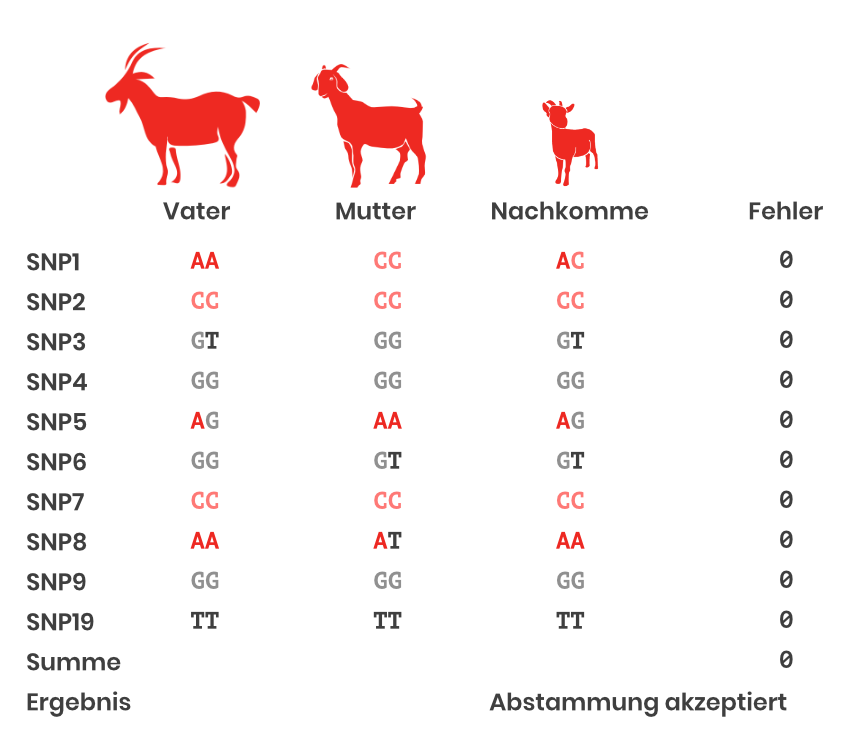
Abbildung 1: Abstammungskontrolle mit SNP (eigene Abbildung).
Nutzen für den Züchter
Ein wesentlicher Unterschied zum bisherigen System ist auch, dass ab sofort die Daten aus der Untersuchung dem SZZV zur Verfügung stehen. Im bisherigen System wurde lediglich das Ergebnis aus der Abstammungskontrolle «Abstammung akzeptiert / abgelehnt» festgehalten und dem Züchter zurückgemeldet.
An der Rückmeldung zum Tierbesitzer ändert sich nichts, diese beschränkt sich auch in Zukunft auf die Kernaussage «Abstammung akzeptiert / abgelehnt«. Zukünftig werden allerdings die 60’000 Genotypen jeder untersuchten Ziege gespeichert und stehen so auch für zukünftige Anwendungen zur Verfügung.
Eine erste unmittelbare Anwendung für den Züchter findet man in der Suche nach möglichen Eltern. Sollte die angegebene Abstammung abgelehnt werden, wird automatisch nach alternativen Eltern gesucht. Die Typisierung von knapp 60’000 SNPs eignet sich also auch für die Suche nach möglichen Eltern und zur Lösung von Problemfällen, wie zum Beispiel mehrere Böcke in einer Herde.
Die Suche nach möglichen Eltern erfolgt in umgekehrter Weise zur Abstammungskontrolle. Unter allen genotypisierten Tieren werden unter Verwendung aller SNPs mögliche Eltern gesucht und ausgewiesen. Diese Suche ist hoch präzise und zuverlässig. Im bisherigen System «Mikrosatelliten» war dies nicht möglich.
SNP – Single Nucleotide Polymorphism
Definition
Der Begriff SNP kommt aus der englischen Sprache und bedeutet «Einzelbasenaustausch». SNPs finden wir im Erbgut der Ziege. Dieses umfasst insgesamt rund 3 Mrd. Basen, die sogenannten genetischen Buchstaben. Es ist in fast jeder Zelle des Organismus vorhanden. Wegen der enormen Grösse braucht es Struktur und Organisation. Zwei wesentliche Grundstrukturen werden unterschieden: Chromosomen und Doppelhelix.
Chromosomen sind Untereinheiten des Erbguts. Die Ziege hat davon 30, wobei eine Ziege jedes dieser Chromosomen einmal vom Vater und einmal von der Mutter geerbt hat. Die Anzahl der Chromosomen ist eine Eigenschaft der Spezies (Abbildung 2).
Jedes dieser Chromosomen besteht aus einer sogenannten Doppelhelix. Dabei sind zwei umeinanderlaufende Stränge gegenseitig verwunden. Jeder Strang ist eine Abfolge der genetischen Buchstaben (A, C, G und T), die aneinander gekettet, den Strang bilden.
Ein Grossteil dieser Stränge ist zwischen verschiedenen Tieren identisch, nämlich rund 99%. Zu den verbleibenden 1% zählen auch unter anderem die SNPs (Abbildung 3). Bei einem SNP trägt ein Tier an dieser Stelle zum Beispiel ein A und ein anderes Tier ein C. Interessant für den Tierzüchter sind die Stellen, an denen auf den Strängen Unterschiede zwischen den Tieren vorhanden sind.
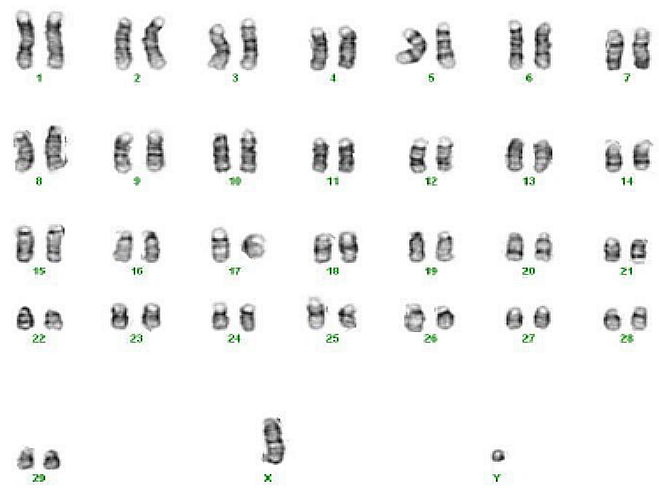
Abbildung 2: Chromosomensatz einer Ziegenbocks (Karyotyp).
Bildquelle: Hadj S Aoued and Mahipal Singh, Wikipedia.
Entstehung
SNPs entstehen spontan im Erbgut und oft bei der Vermehrung des Erbgutes. Je nach Position des SNP und der Zelle, in der es entsteht, kann die Wirkung eines neuen SNP sehr unterschiedlich sein. SNPs, welche spontan zum Beispiel in einer Körperzelle entstehen, werden nicht an die Nachkommen weitergegeben, sondern sind beschränkt in ihrem Auftreten auf die jeweilige Zelle sowie deren Tochterzellen aus der Zellteilung. Hingegen werden SNPs, die in der Keimbahn, den Samen- bzw. Eizellen, entstehen, über die Generationen hinweg vererbt.
Durch das Verhalten der Züchter und die Definition beziehungsweise Trennung von Rassen ist es somit durchaus möglich, dass ein SNP in seinem Auftreten auf eine oder wenige Rassen beschränkt ist.
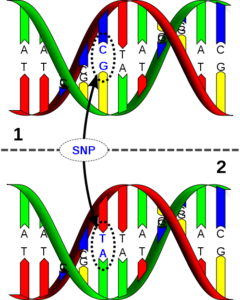
Abbildung 3: Einzelbasenaustausch (SNP).
Bildquelle: Wikimedia.
Wirkung
Je nach Position eines SNP (innerhalb oder ausserhalb eines Gens) sind seine funktionellen Auswirkungen sehr unterschiedlich. Längst nicht jeder SNP hat zwangsläufig eine (heute bekannte) Funktion. So ist die unmittelbare Funktionalität nur von einem kleinen Bruchteil aller SNPs heute überhaupt bekannt. Über den weitaus grössten Teil der SNP ist auch heute nichts beziehungsweise wenig von ihrer Wirkung bekannt.
Zu den SNPs mit bekannten Auswirkungen gehören zum Beispiel SNPs in Farbgenen oder Milchproteingenen. Diese SNPs führen zur Ausprägung kategorischer Merkmale wie Eiweisstyp oder Farbe.
Noch schwieriger wird der Nachweis eines SNP auf ein quantitatives Merkmal. Dennoch gibt es auch hier ein Paradebeispiel: der Effekt von einem SNP aus dem Gen namens DGAT1 auf das Merkmal Fettgehalt. Tiere, die an dieser Stelle im Genom ein C aufweisen, haben einen deutlich höheren Fettgehalt in der Milch als Tiere mit einem T an entsprechender Position.
Typisierung
Im Verlauf der letzten Jahrzehnte entwickelten sich verschiedene Technologien für die Analyse, welche Buchstaben in einem SNP eines Tieres vorhanden sind. Diese Analyse nennt man Typisierung.
Der Fortschritt in diesem Bereich war enorm. Angetrieben vom Motor der Grundlagenforschung sind seit inzwischen mehr als zehn Jahren SNP-Chips auch für Nutztierarten verfügbar. Das bekannteste Beispiel für die Anwendung dürfte die genomische Selektion beim Rind sein, wo seit 2010 SNP-Chips nicht mehr wegzudenken sind.
SNP-Chips sind vereinfacht ausgedrückt Einheiten (Arrays), bei denen man im Hochdurchsatz pro Tier mehrere tausend SNP gleichzeitig untersucht. Der technologische Fortschritt bewirkte auch einen massiven Rückgang der Kosten. Umgerechnet kostet daher eine Typisierung heute weniger als 0.1 Rappen pro SNP. Damit wurde eine praktische Anwendung ausserhalb von Forschungsprojekten überhaupt erst möglich.
Chips
Wie eingangs erwähnt, muss man davon ausgehen, dass im Ziegengenom rund 30 Mio. SNPs vorhanden sind. Dies übersteigt die Möglichkeiten eines Chips, da mit dieser Technologie maximal rund 1 Mio. SNPs analysiert werden können. So muss aus dem theoretisch vorhandenen SNP-Pool für die Chip-Typisierung eine Auswahl getroffen werden.
Eine Schlüsseleigenschaft ist die gegenseitige Lage der SNP, da mit einer gleichmässigen Verteilung der ausgewählten SNP ein Netz an Markern über das Erbgut gelegt wird. Dieses Netz ist entscheidend für die folgenden Anwendungsmöglichkeiten, unter anderem die genomische Selektion.
Für ein möglichst breites Anwendungsspektrum eines Chips sollten auch SNPs für die Abstammungskontrolle enthalten sein. Diese müssen nämlich eine zusätzliche Eigenschaft erfüllen und in möglichst vielen verschiedenen Rassen vorhanden sein, damit die Abstammungskontrolle mit demselben Chip für sämtliche Ziegenrassen durchgeführt werden können.
Aktuell sind auf dem Chip 195 SNPs für die Abstammungskontrolle enthalten. Relevante Zusatztests zum Beispiel für Kappa Kasein (Milchprotein) runden einen Chip ab. Der Chip, der für die Abstammungskontrolle verwendet werden wird, ist eine Erweiterung des seit 2014 bestehenden Chips. Vor allem im Bereich der Zusatztests wurde der Chip erweitert. Er wurde durch ein internationales Konsortium entwickelt, bei dem auch Schweizer Wissenschaftler des Instituts für Genetik der Universität Bern einen Beitrag geleistet haben.

(eigene Abbildung)
Ausblick
Im Vordergrund steht die Einführung der Abstammungskontrolle. Da aber im Chip eine Reihe von zusätzlichen Markern (Kappa Kasein, DGAT1) enthalten sind, wird deren Verarbeitung und Publikation im Laufe des Jahres folgen.
Über das internationale Projekt SMARTER, in dem ebenfalls Genotypen von Schweizer Ziegen generiert werden, wird sich die Datenmenge schnell vergrössern, so dass theoretisch auch weiterführendere Anwendungen (genomische Selektion) in Reichweite gelangen.
Es ist wünschenswert, wenn die Züchter in ihrem Alltag zukünftig möglichst zahlreich von der SNP-Typisierung Gebrauch machen, da dies der Ziegenzucht schnell zu Gute kommt.


