Dans cet article du blog, vous découvrirez le développement de la sélection génomique de la Limousine pour le caractère poids au sevrage direct qui sera introduit le 7 avril 2020.

Connaissances de base sur la sélection génomique
La sélection génomique travaille sur la base de marqueurs SNP (single nucleotide polymorphism) qui sont répartis plus ou moins uniformément sur le patrimoine génétique de l’animal. Les SNP sont des changement d’une molécule à une position précise du génome. En ce moment, deux puces sont couramment utilisées pour génotyper les animaux. Elles contiennent environ soit 150’000 SNP (Bovine150k-Chip) ou 45’000 SNP (LD-Chip). Par la répartition uniforme de ces nombreuses SNP sur le génome, on part du principe que des SNP se trouvent à proximité de gènes qui ont une influence sur des caractéristiques importantes pour le travail de sélection fait par les éleveurs.
L’ampleur de l’effet de chaque marqueur SNP doit être estimée. A ce fin, un lien doit être fait entre les génotypes (les versions de marqueur) et les performances des animaux (les valeurs d’élevage conventionnelles). L’estimation de l’effet de chaque marqueur est basée sur les génotypes et performances des animaux qui ont des valeurs d’élevage de haute sécurité. Ces animaux sont considérés alors comme une population de référence. Plus il y a d’animaux avec des valeurs d’élevage sécurisées à disposition, plus l’estimation de l’effet de chaque marqueur SNP peut être précise. La population de référence actuelle pour le poids au sevrage direct (PSD) compte 3’593 animaux en avril 2020.
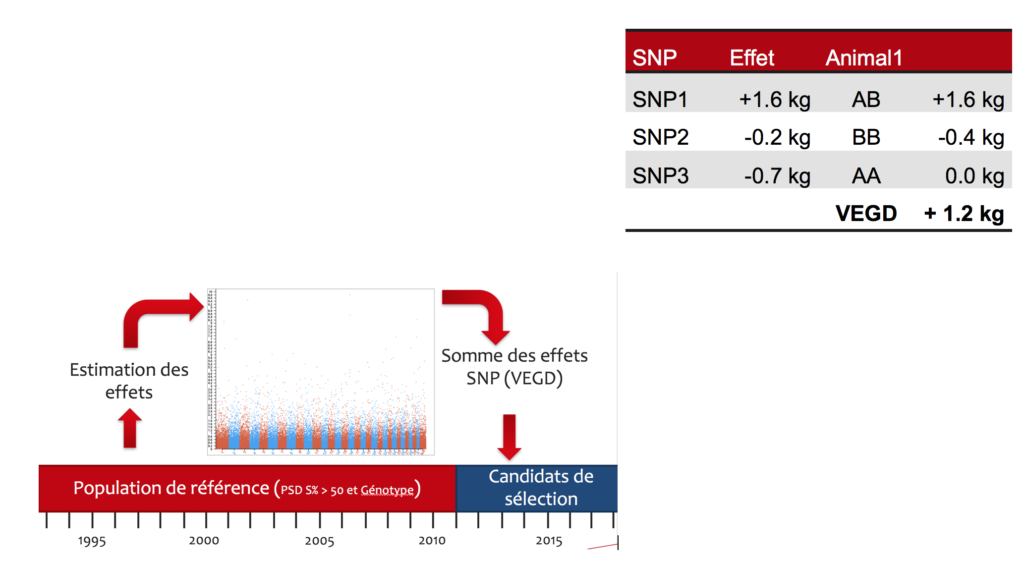
Illustration 1: Estimation des effets à l’aide de la population de référence et calcul de VEGD pour les candidats de la sélection.
L’addition de l’effet de la variante de chaque marqueur que porte un animal permet de calculer sa valeur d’élevage génomique directe (VEGD). Une fois l’estimation de l’effet de chaque marqueur faite, tout animal génotypé de la même race que les animaux de la population de référence peut donc avoir une valeur d’élevage génomique directe même s’il n’a pas encore de valeurs d’élevage conventionnelles. (Illustration 1)
La sécurité des valeurs génomiques est estimée suivant la correspondance entre les VEDG et les valeurs d’élevage conventionnelles des animaux de validation. Pour ce faire, 10% des animaux les plus jeunes de la population de référence sont exclus lors de l’estimation des effets des marqueurs SNP. Une fois les effets des marqueurs estimés avec les 90% des animaux de référence restant, les VEDG des animaux de validation sont comparé avec leur valeurs d’élevage traditionnelles. En effet, la sécurité des VEDG de tous les animaux génotypés est la corrélation entre les VEDG et les valeurs d’élevage conventionnelles des animaux de validation.
La meilleure estimation du potentiel génétique d’un animal est possible lorsque toutes les informations disponibles (conventionnelles et génomiques) sont utilisées et résumées dans une valeur combinée. Cette valeur combinée se nomme la valeur d’élevage génomique optimisée (VEGO). Le poids de la valeur d’élevage génomique et conventionnelle dans la VEGO est pondéré par la sécurité de ces deux valeurs d’élevage. Pour des jeunes animaux avec une valeur d’ascendance (sécurité basse), la pondération de la VEGD est plus élevée. Dès qu’un animal obtient des performances propres ou de ses descendants, la pondération de la valeur d’élevage conventionnelle augmente et la pondération de la VEDG devient moindre dans la VEGO. Dans le cas des animaux avec une sécurité de valeur d’élevage conventionnelle très élevée, la VEGO correspond à cette dernière.
Région qui est impliquée dans l'expression de PSD
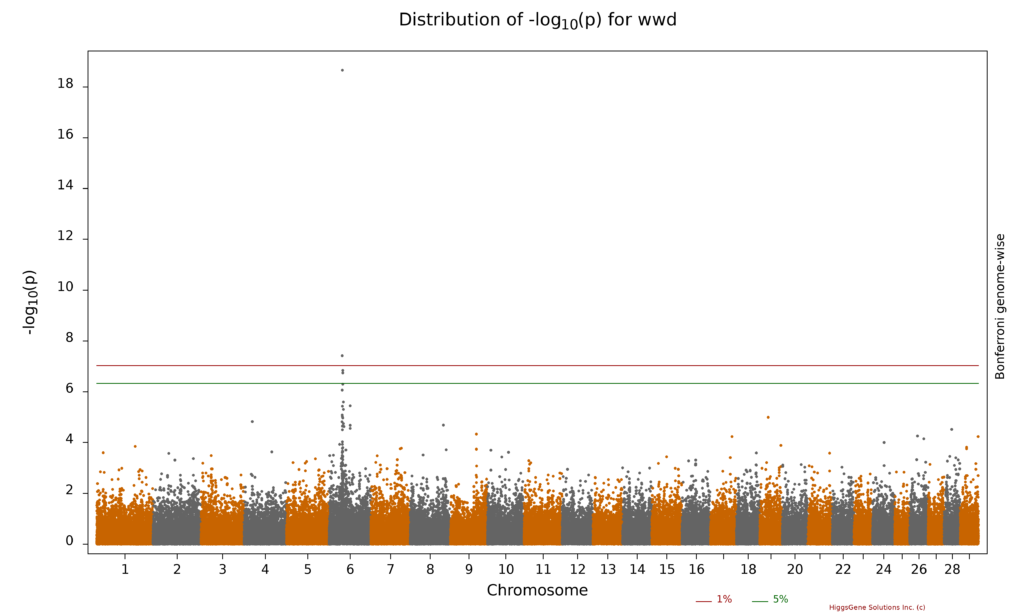
Illustration 2: Région du chromosome 6 de la race Limousine qui est impliquée dans l’expression de PSD.
Lors du développement de la sélection génomique pour le PSD dans la population Limousine, une région du génome, dit QTL (quantitative trait loci), a pu être localisée avec les marqueurs SNP. Cette région est impliquée dans l’expression de PSD et se trouve sur le chromosome 6 (Illustration 2).
Dans un article scientifique, Saatchi et al (2014) ont démontré que cette région peut être associée à plusieurs caractères des races à viande. Nos propres données, nous ont permis de confirmer que cette région à un impact sur le PSD dans la population Limousine génotypée en Suisse.
Mise en place de la sélection génomique
Les valeurs génomiques pour PSD seront publiées le 7 avril 2020 pour la première fois. Dès lors, Vache Mère Suisse procèdera à trois évaluations génétiques de routine (les dates de publication 2020: 7 Avril / 11 Août / 1 Décembre). Lors de ces évaluations génétiques de routine, l’estimation des effets pour la sélection génomique est actualisée (selon le descriptif des connaissances de base et l’Illustration 1).
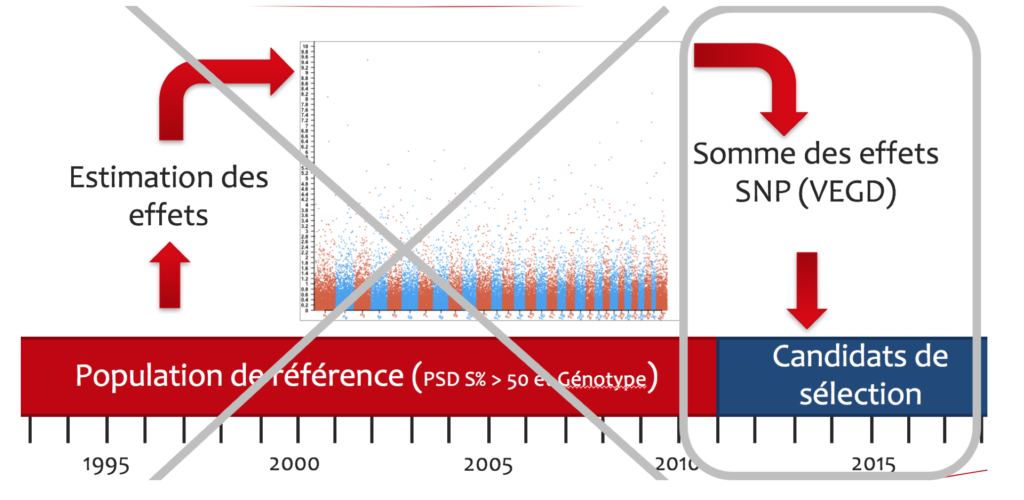
En plus de ces trois dates de publication, tous les deuxièmes mardi, les VEDG et VEGO des animaux nouvellement génotypés (p.ex. veaux) seront estimées en se basant sur les effets estimés pour la dernière publication officielle des valeurs d’élevage en date (Illustration 3). De cette manière, les éleveurs peuvent envoyer leur échantillons pour génotypage auprès de Qualitas et recevront les résultats de leur animaux en continu par mail. Il faut compter environ un mois entre l’envoi de l’échantillon et l’arrivée des résultats d’analyse.
La différence entre les trois publications de routine et les publications des mardis, est que lors des publications des mardis l’estimation des effets n’est pas actualisée et se base donc sur la dernière évaluation génétique de routine. Trois fois par année après l’estimation des valeurs d’élevage conventionnelles, les VEGO de tous les animaux sont à nouveau estimées et publiées.
Par l’utilisation de la technologie basée sur les SNP, à côté de la sélection génomique, il y a d’autres synergies:
Contrôle d’ascendance – Sélection génomique – Test de gènes spécifiques
Est-ce qu'il y a d'autres caractères dans la pipeline?
Par la même occasion que pour le développement de la sélection génomique pour le caractère PSD, l’application de la sélection génomique au reste des caractères pour la Limousine a été étudié. Malheureusement et contre toute attente, aucun gain en information n’a pu être confirmé avec la sélection génomique pour le moment. Nous travaillons encore sur ce sujet en mettant un accent principal sur les caractères de la carcasse et de son évaluation génétique conventionnelle. Des changements sont attendus dans l’évaluation génétique de ces caractères et nous vous informerons à ce sujet en temps voulu.
Quelques approches sur lesquelles nous travaillons:
- Une définition des caractères plus fine et une différenciation des systèmes de production de vache allaitantes (veaux qui têtent la mère) ou d’engraissement conventionnel (veaux qui ne têtent pas la mère) et une différenciation des veaux d’étal (Natura-Veal / tous les autres veaux d’engraissement conventionnel) et du bétail d’étal (Natura-Beef / SwissPrimBeef / tous les autres bêtes d’engraissement conventionnel)
- Considération de l’âge à l’abattage en tant que caractère au lieu de covariable comme jusqu’à présent dans le modèle
- Meilleure représentation de l’effet de différentes races sur les performances des animaux croisés
- etc.
Une fois l’optimisation de l’évaluation génétique conventionnelle des carcasses terminée, le modèle pour l’évaluation génétique des naissances sera révisé.
Résumé
Le caractère PSD correspond à la croissance du veau. Avec l’introduction de la sélection génomique pour ce caractère au sein de la race Limousine, les éleveurs ont la possibilité de sélectionner les animaux avec les meilleures disposition génétique pour ce caractère plus tôt dans la vie des animaux.


