Das Thema Methodik in der genomischen ZWS war auch an dieser Konferenz sehr prominent vertreten. Bereits in der ersten Plenary session wurde von Mario Calus aufgezeigt, dass dieses Jahr erstmals mehr Paper zu Genomic prediction als zu GWAS und QTL prediction eingereicht wurden.
Zum einen gab es Sessions die spezifisch nur dem Thema «Methods and Tools» gewidmet waren. Zum anderen wurde der Themenkreis “genomische Selektion”, insbesondere die Single-Step-ZWS, auch immer wieder bei anderen Themen aufgegriffen und diskutiert.
Als erstes Beispiel wurde von Daniela Lourenco von der University of Georgia die Weiterentwicklung der blupf90-Programme vorgestellt. Dieses bekannte Programmpaket wurde grundlegend überarbeitet. Es kann vor allem für Varianzkomponentenschätzung und die traditionelle und genomische ZWS eingesetzt werden. Mit ihm können aber auch viele vor- und nachgelagerte Prozesse, die für die oben genannten Methoden wichtig sind, durchgeführt werden.
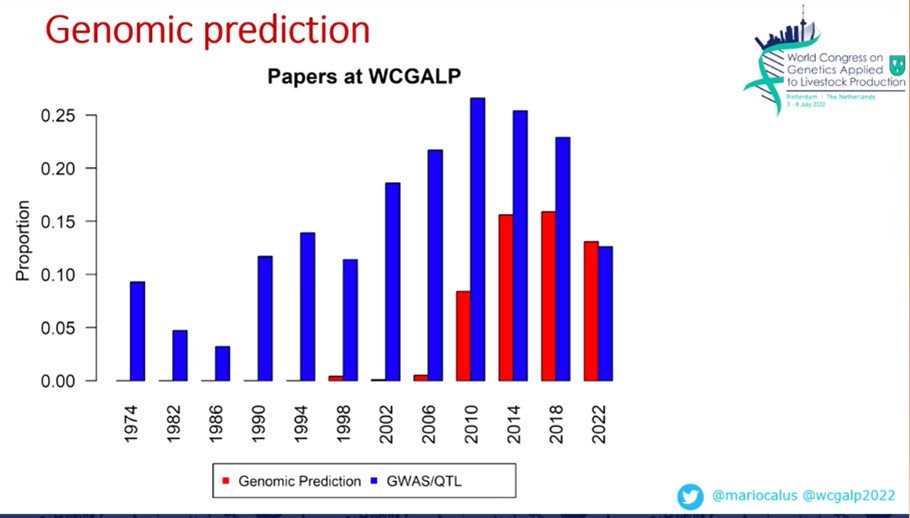
Abb 1: M.Calus WCGALP 2022 Anzahl eingereichter Papers zu den Themen GWAS/QTL (blau) und Genomic Prediction (rot)

Abb 2: Frischknecht et al. 2016, EAAP Belfast : Genauigkeit der genomichen ZWS mit verschiedenen Markerdichten im Vergleich zur 50k Dichte für die Merkmale NRH: non-return rate Rind, SCS: somatic cell score und STA: Kreuzbeinhöhe
In der Session “Challenges – Use of whole genome sequence information” wurden verschiedene Beiträge gezeigt, wie die Auswahl von Markern für die genomische Selektion optimiert werden kann. Wir haben bereits vor einigen Jahren dargelegt, dass es wenig Genauigkeitsgewinn bringt, wenn sämtliche imputierten Sequenzvarianten berücksichtigt werden. Diese Grunderkenntnis haben verschiedene Vorträge in dieser Session bestätigt und neue Verfahren zur Auswahl eines optimalen Markersets vorgestellt.





