Die Plenary Session am Freitagvormittag war ganz dem Thema genomweite Assoziationsstudien gewidmet. Hier wurde eine der weltweit grössten genomweiten Assoziationsstudien präsentiert: In diese Studie flossen die Grössenmessungen und Genotypen von knapp 5 Millionen Menschen ein. Loic Yengo von der University of Queensland in Brisbane, Australien, hat diese Studie vorgestellt . Er hat interessante Aspekte der Vererbung der menschlichen Grösse aufgezeigt, aber auch auf die Schwierigkeiten solch riesiger Datensätze hingewiesen. Insbesondere im Humanbereich werden oft nicht Rohdaten, sondern nur sogenannte «summary statistics» geteilt und daher geht auch ein Teil der Informationen verloren. Im Rinderbereich werden einige Länder wohl bald eigene Datensätze in solchen Grössenordnungen haben.
Genetische Marker müssen nicht zwangsläufig über einen kommerziell erhältlichen Chip typisiert werden. Bei der Qualitas ist seit einigen Jahren ein sogenannter «custom-chip» im Einsatz. Die Varianten, die auf diesem Chip typisiert werden, wurden von Genetiker*innen der Qualitas in einem Projekt gemeinsam mit der Universität Bern ausgewählt und in Zusammenarbeit mit dem Labor und dem Chiphersteller entworfen und validiert. Auch andere gehen den Weg eines custom-chips. So wurde beispielsweise für das Niederungsrind, eine alte Rinderrasse aus Deutschland, ein eigener Chip entwickelt, bei dem populationsspezifische Marker berücksichtigt wurden.
Ein anderer Ansatz, um die Genotypen zu erfassen, ist die Methodik “low-pass-sequencing“, eine vielversprechende Technologie, die in den letzten Jahren aufgekommen ist und seither weiter verfeinert wird. Bei dieser Technologie werden die Tiere genomweit sequenziert, aber nur mit einer sehr geringen Abdeckung. Fehlende Marker werden anschliessend mit einem Referenzpanel imputiert. Das Ziel ist, eine viel höhere Markerdichte als mit Chips zu erreichen.

Abb.: Fleischrinder (Bildquelle: Karl Sulz)
Weitere Vorträge gingen mehr auf einzelne genomische Regionen ein. So zeigte Troy Rowan einen alternativen Ansatz für die Identifikation von Selektionssignaturen: Es wird das Geburtsjahr des Tieres als Näherung für die Generation als Phänotyp genommen und eine genomweite Assoziationsstudie durchgeführt. Dies wurde für verschiedene nordamerikanische Fleischrinderrassen gemacht und interessante Ergebnisse erzielt.
Auch Aurelien Capitan, von der INRAE in Frankreich, hat in seinem Vortrag das Thema der letalen Haplotypen bei Rindern erweitert, so dass nun auch Haplotypen mit inkompletter Penetranz identifiziert werden können. Dazu wurden die Haplotypen in verschiedenen Tiergruppen verfolgt. Kamen die Haplotypen öfter in Gruppen vor, bei denen mehr Tiere starben, war dies ein Hinweis für einen solchen Haplotypen mit unvollständiger Penetranz. Es wurden dazu bereits erste Varianten präsentiert. In der Abbildung ist ein Beispiel aufgezeigt, wie Tiere, die Haplotypenträger sind, überleben.
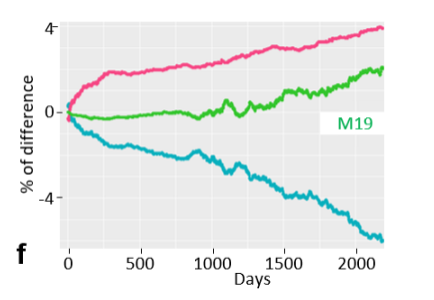
Abb.: Unterschied in der Abgangsrate von Tieren mit verschiedenen Trägerstati des Haplotypen M19. Rot: Gestorben; Grün: Geschlachtet; Blau: Lebend. Bei diesem Haplotypen haben Trägertiere über die gesamt eAufzuchtperiode ein erhöhtes Abgangsrisiko. (Bildquelle: Guintard, A. et al. 2022 Massive detection of cryptic recessive genetic defects in livestock mining millions of life trajectories., WCGALP)



